LigandScout教程(三): 基于配体的药效团建模
一、操作步骤
- 导入配体
- 生成3D结构,对配体聚类
- 生成基于配体的药效团模型
二、详细描述
1,导入配体,设置训练集化合物
假设已知活性化合物以SMILES格式保存在cdk2.smi文件里。
File>>Open>>cdk2.smi
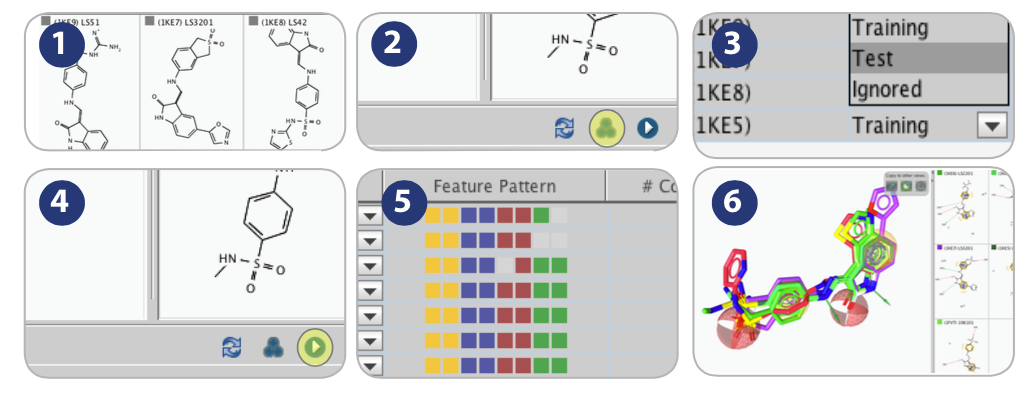
配体会被导入,其2D结构展示在屏幕的右侧。[见图示1]
2,设置训练集化合物
将用来建立模型的化合物选中,设置选中的化合物为训练集化合物。
Ligand-Set>>Flag Selected Molecules as Training Set
3, 根据3D药效团特征对训练集进行聚类
点击“Cluster”按钮[图示2],用默认值,再点击OK按钮。
配体表单会出现一个新的列:Cluster ID。点击Cluster id,进行排序。
4,设置测试集化合物
通过表单控制,将cluser id为1的化合物设为“test set”。[图示3]
5,建立基于配体的药效团模型
点击“Create Ligand-based pharmacophore”按钮[图示4],生成药效团模型。
“Feature Pattern”展示了药效团元素组成[图示5],这些药效团元素与所有的化合物都匹配。点击一个色块,会链接向对应的药效团元素。在右上角展示了生成的一组药效团模型:点击不同模型,对应的3D模型与配体就会屏幕上展示出来。[图示6]
三、从这里开始,我能做些什么?
- 将基于结构的模型与基于配体的模型进行叠合
- 在LigandScout里对数据库进行虚拟筛选
四、应用案例
Anna Vuorinen et al. Ligand-Based Pharmacophore Modeling and Virtual Screening for the Discovery of Novel 17β -Hydroxysteroid Dehydrogenase 2 Inhibitors. J. Med. Chem. 2014, 57, 5995 −6007
阅读:http://www.molcalx.com.cn/ligandscout_jmc2014
五,联系我们,获取一个月的免费试用
[contact-form-7 id=”832″ title=”contact”]