健壮、高效的计算
- 无缝的蛋白与配体准备
- 用Flare的配体叠合工具可以轻松的将化合物叠合好,或者用Flare的分子对接可以轻松的将配体叠合到FEP项目相关的蛋白结合位点里
- 全自动、高效地创建FEP网络,提供选项优化映射
- 在专用的视窗中轻松地查看结果、问题排除、识别有问题的转化
- 用预测的活性对配体进行可视化分析、排序与优先性排序
精确的FEP计算
- 对一系列结构紧密相关的配体(或同系物)用炼金术法精确地计算相对结合自由能(参见J. Chem. Inf. Model. 2020, 60(6):3120–3130)
- 在Flare FEP计算中使用完全连接的双向网络可获得精确度,从而可以更准确地估计每个链接的误差并彻底分析循环误差。
- 通过中间体的自动添加可以更好地链接结构相关的配体,轻松生成网络图
- 全自动、高效地创建FEP网络,提供选项优化映射
- 在专用的视窗中轻松地查看结果、问题排除、识别有问题的转化
- 用预测的活性对配体进行可视化分析、排序与优先性排序
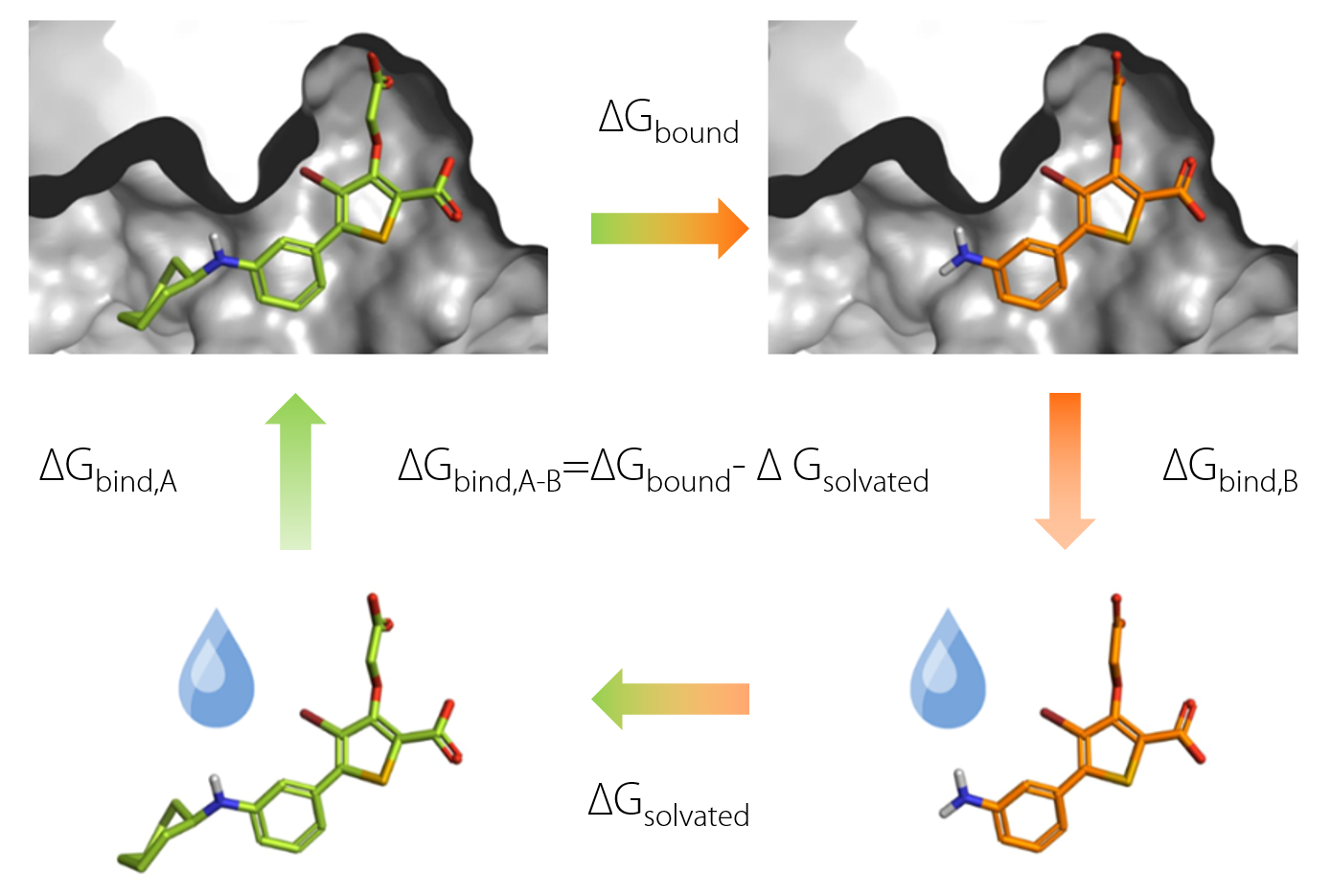
自由能循环
快速的FEP计算
- 将您的 Flare FEP 计算拆分为适合您 GPU 资源的最佳包大小,从而在短短 30 分钟内预测单个化合物
- Flare FEP的模拟计算使用了先进的积分器、氢质量重新分配和优化的水盒形状和尺寸
- 通过使用Cresset Engine Broker连接到本地集群或云计算设施来优化计算时间
- 由软件自适应地自动确定每个微扰之间λ 窗口数量的分配,实现量身定制、更快速的模拟
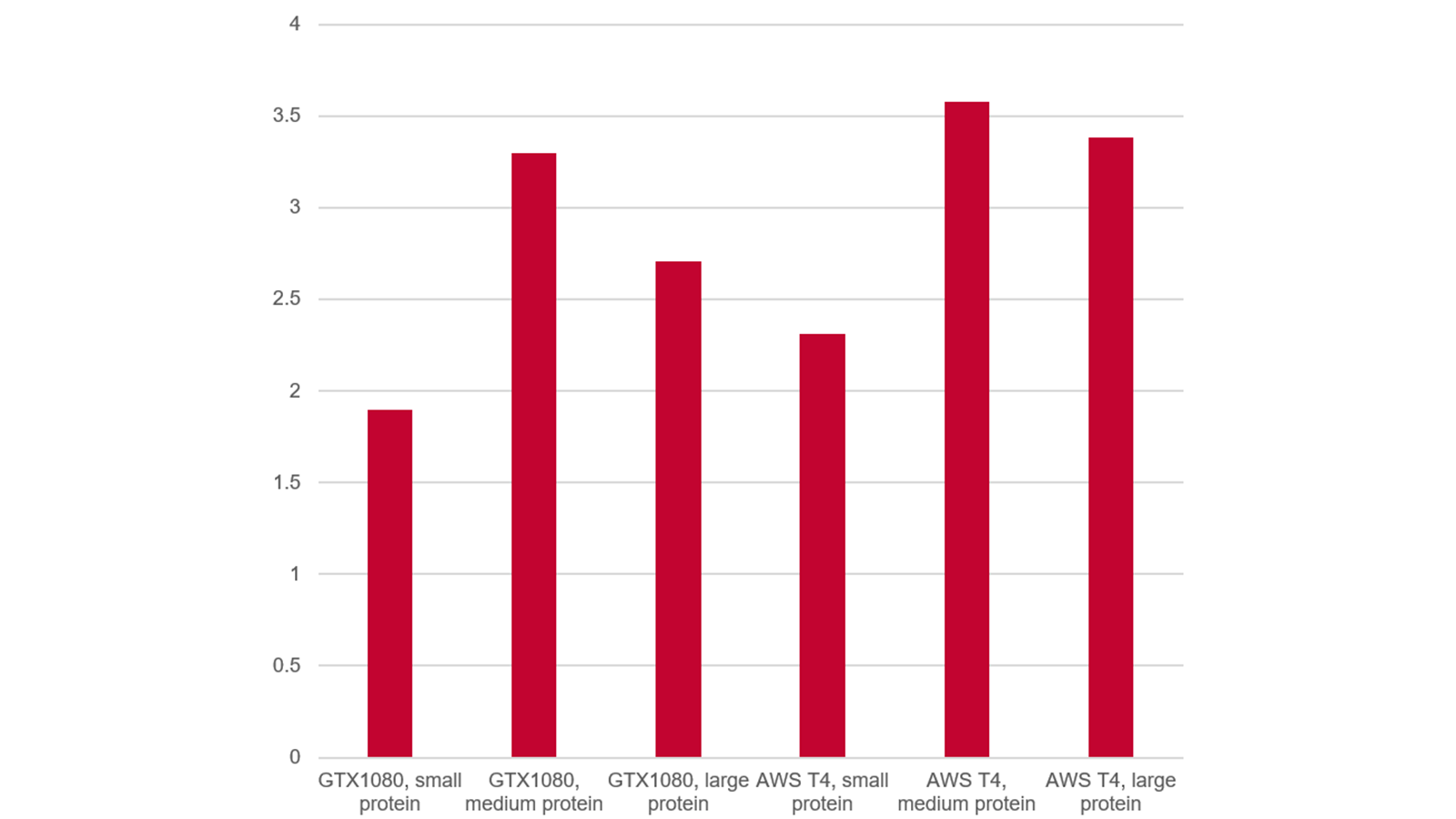
比之Flare V4, Flare V5的FEP计算速度提高了2-3倍
用云计算加速您的药物发现
为了加速您的药物发现,与国内超算中心与云服务商合作,确保您可以在没有自己集群或自有资源不足的情况下利用云计算安全、高效进行FEP计算。我们已经在AWS、百度云、腾讯云、速石科技、北京超级云计算中心以及北琨云等平台上部署测试过。下面为大家介绍一些测试案例,为大家提供参考。
- 用云资源加速药物发现——Flare FEP在北京超级云计算中心GPU资源上的性能测试
- 用云资源加速药物发现——速石科技GPU资源进行FEP计算的性能表现
- FLARE算例 | 炼金术法FEP预测FXR激动剂的结合自由能
推荐阅读
参考文献与致谢
- P. Eastman, J. Swails, J. D. Chodera, R. T. McGibbon, Y. Zhao, K. A. Beauchamp, L.-P. Wang, A. C. Simmonett, M. P. Harrigan, C. D. Stern, R. P. Wiewiora, B. R. Brooks, and V. S. Pande, OpenMM 7: Rapid development of high performance algorithms for molecular dynamics, PLOS Comp. Biol. 2017, 3(7): e1005659
- S. Liu, Y. Wu, T. Lin, R. Abel, J. P. Redmann, C. M. Summa, V. R. Jaber, N. M. Lim, D. L. Mobley, Lead optimization mapper: automating free energy calculations for lead optimization, J. Comput. Aided Mol. Des. 2013, 27, 9, 755-770
- L. O. Hedges, A. S. J. S Mey, , C. A. Laughton, F. L. Gervasio, A. J. Mulholland,C. J. Woods, J. Michel, BioSimSpace: An interoperable Python framework for biomolecular simulation, Journal of Open Source Software 2019, 4(43), 1831
- C. Woods , L. O. Hedges, A. S. J. S. Mey , G. Calabrò and J. Michel , www.siremol.org, Sire Molecular Simulation Framework , 2022
- G. Calabrò , C. J. Woods , F. Powlesland , A. S. J. S. Mey , A. J. Mulholland and J. Michel , Elucidation of Nonadditive Effects in Protein–Ligand Binding Energies: Thrombin as a Case Study, J. Phys. Chem. B, 2016, 120 , 5340 —5350
- H. H. Loeffler, J. Michel, C. J. Woods, FESetup: Automating Setup for Alchemical Free Energy Simulations, J. Chem. Inf Model. 2015, 55 (12), 2485-2490
- C. Devereux, J. S. Smith, K. K. Huddleston, K. Barros, R. Zubatyuk, O. Isayev, and A. E. Roitberg, Extending the applicability of the ANI deep learning molecular potential to sulfur and halogens, J. Chem. Theory Comput. 2020, 16,7, 4192–4202
Flare™
进一步探索Flare如何为你的分子设计增加新的见解