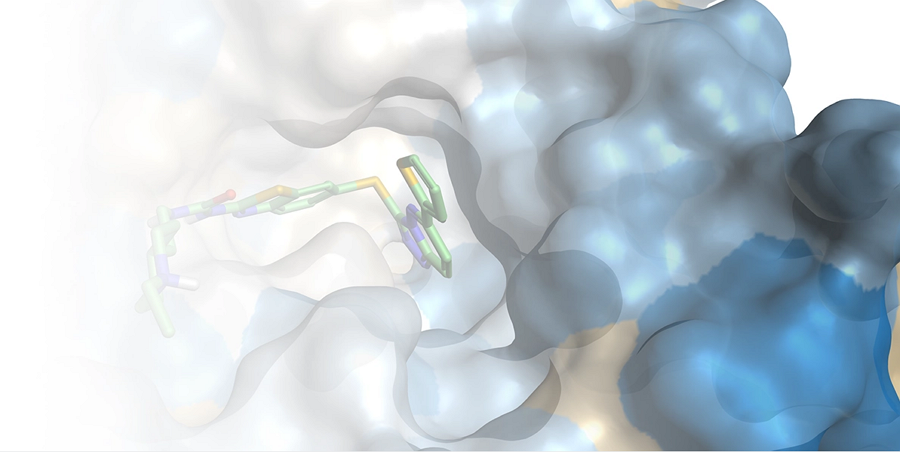
蛋白构象的动态稳定性
蛋白质构象的动态稳定性已成为研究蛋白质和蛋白质-配体复合物的关键技术。Flare™提供了一个专用界面来创建、回放和分析分子动力学模拟的轨迹。分子动力学模拟轨迹分析可以帮您:
- 通过接触频次分析以确认对接结合模式的稳定性。
- 在轨迹回放期间查看初始配体的结合模式,以直观地评估配体运动以及结合模式变化。
- 轻松截取快照作为进一步实验的起点,例如系综对接或FEP计算。

定制和快速设置实验
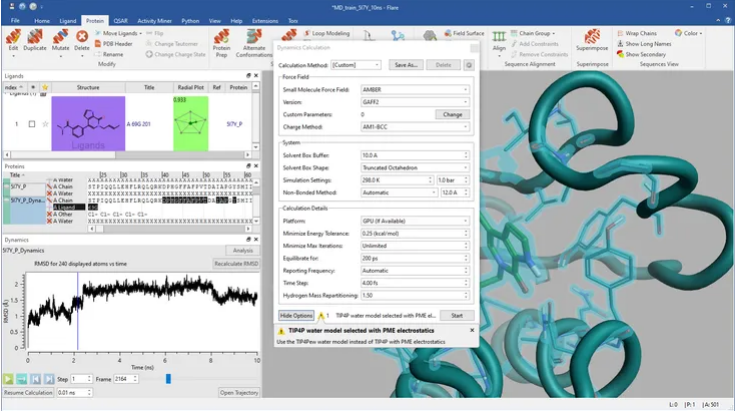
Flare支持深度定制或标准的计算配置,从而实现定制、快速的实验设置。分子动力学模拟计算的主要特点是:
- 支持多种力场,包括:AMBER GAFF/GAFF2或者是新型的OpenForceField。
- 四点显式水模型可供选择以获得最大的精度。
- 多种隐式溶剂模型可供选择实现快速模拟计算。
- 使用氢质量再分配和更长的时间步长实现在更短的时间内进行更长的模拟计算。
- 提供力场参数构建器为您的分子创建自定义参数,确保结果的准确性。
- 支持本地GPU/CPU 或通过Cresset Engine Broker无缝连接到远程计算资源来加速计算。
- 多种隐式溶剂模型可供选择实现快速模拟计算。
在Flare中分析分子动力学模拟的轨迹
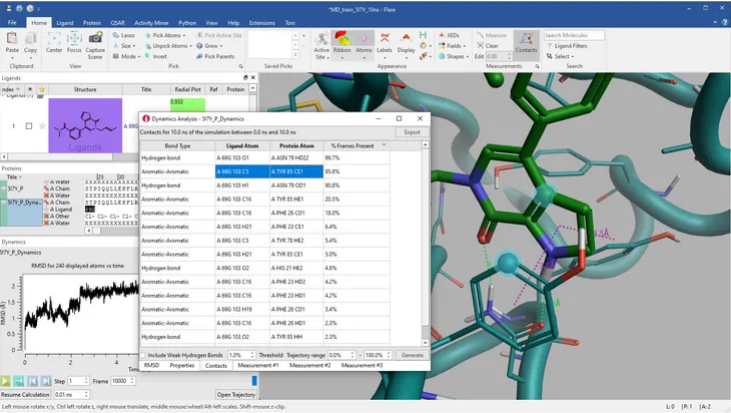
在Flare中分析分子动力学模拟轨迹为您提供决策所需的反馈。
- 专用的分子动力学模拟界面可以在完整Flare项目的环境中可视化轨迹,从而可以与起始蛋白-配体构象进行比较。
- 四点显式水模型可供选择以获得最大的精度。
- 通过分析系统的势能、温度、盒子体积以及密度等系统物理特性,获得对结果的信心。
- Flare使用强大的OpenMM作为计算引擎,可以在任何计算轨迹的地方以标准DCD格式保存和读取轨迹。
案例分析
参考文献与致谢
- P. Eastman, J. Swails, J. D. Chodera, R. T. McGibbon, Y. Zhao, K. A. Beauchamp, L.-P. Wang, A. C. Simmonett, M. P. Harrigan, C. D. Stern, R. P. Wiewiora, B. R. Brooks, and V. S. Pande, OpenMM 7: Rapid development of high performance algorithms for molecular dynamics, PLOS Comp. Biol. 2017, 3(7): e1005659
- C. Devereux, J. S. Smith, K. K. Huddleston, K. Barros, R. Zubatyuk, O. Isayev, and A. E. Roitberg, Extending the applicability of the ANI deep learning molecular potential to sulfur and halogens, J. Chem. Theory Comput. 2020, 16,7, 4192–4202
Flare™
进一步探索Flare如何为你的分子设计增加新的见解