LigandScout应用案例第二期
LigandScout应用案例-基于结构的虚拟筛选发现全新DNA促旋酶抑制剂
文献来源
MatjažBrvar, Andrej Perdih, Miha Renko, Gregor Anderluh, Duš an Turk, Tom Solmajer. Structure-Based Discovery of Substituted 4,5’-Bithiazoles as Novel DNA Gyrase Inhibitors. Journal of Medicinal Chemistry. 2012, 55, 6413-6426.
摘要
细菌DNA促旋酶是一种久经检验并得到确认的新型抗菌药的靶标。它对细菌的DNA复制周期起到关键作用,却不存在于哺乳类动物。这使得细菌DNA促旋酶成为了发展具有选择毒性的抗菌药物的良好靶标。DNA促旋酶B亚型的ATP结合位点非常重要,并已有广泛研究。为了发现细菌DNA促旋酶B亚型ATP结合位点的全新抑制剂,作者采用了两步设计策略:第一阶段中使用LigandScout软件分析CBN(一种香豆素类抑制剂)与G24蛋白(包含ATP结合位点的N端最小的片段)之间的相互作用,构建药效团模型进行虚拟筛选,然后用分子对接方法进一步筛选;第二阶段,对第一阶段识别出来的4,5’-二噻唑结构类型构建集中库,采用对接方法进行筛选。抗菌活性测试结果确证了几个化合物具有DNA促旋酶B型的抑制活性,对其中最好的抑制剂还采用SPR、MST、DSF及X-ray等多种实验技术进行结构表征。
计算步骤
阶段一
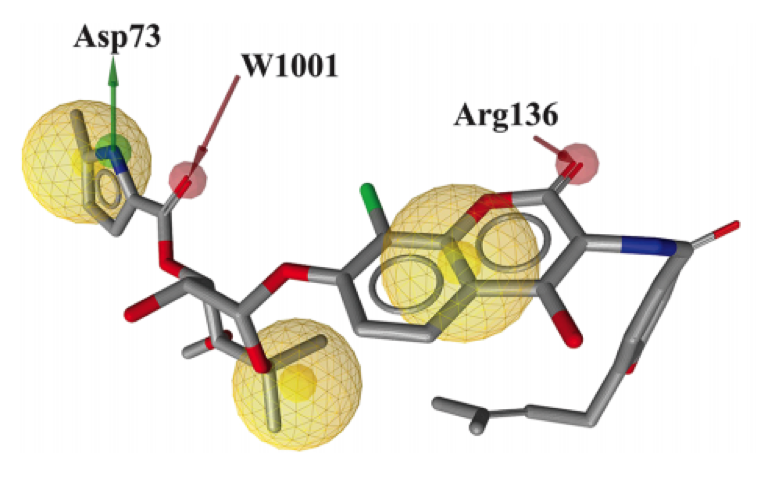
- 用LigandScout基于结构的方法,分析蛋白-配体相互作用,构建并检验药效团模型;
图一、LigandScout产生的简化药效团模型。
- 虚拟筛选
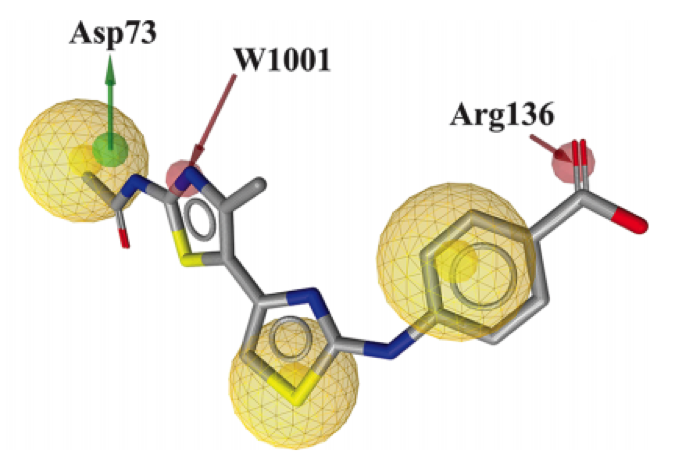
为增加苗头化合物的多样性,对上述药效团模型进行简化和调整,然后对约含5百万个化合物的商业库进行虚拟筛选,命中大约400个不同结构类型的苗头化合物。
图二、虚拟筛选苗头化合物6与药效团模型的叠合。
- 分子对接
采用GOLD进行分子对接计算,将上述苗头化合物对接到DNA促旋酶B活性位点,得到12个不同结构类型的化合物;
- 生物学验证
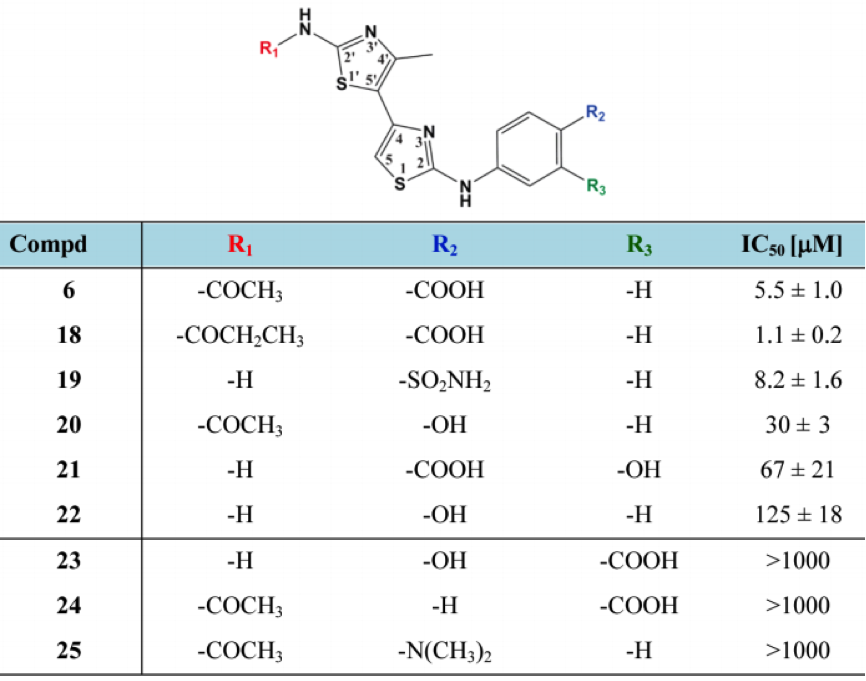
有2个4,5’-二噻唑类化合物具有DNA促旋酶抑制活性,其中化合物6具有低微摩尔活性(IC50 = 5.5 ± 1.0 μM)。
阶段二
- 构建集中库
从可购买化合物的eMolecules数据库中获取240个分子组成的4,5’-二噻唑类化合物集中库,并由OMEGA软件产生3D结构。
- 分子对接
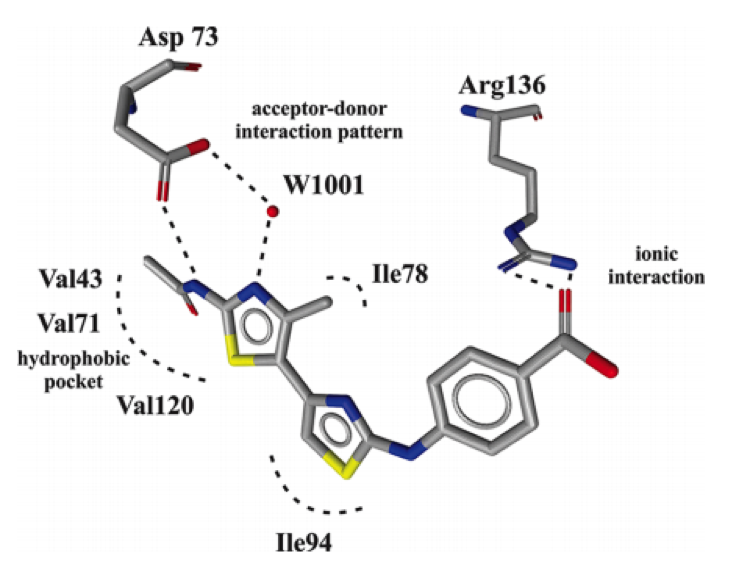
再次采用GOLD进行分子对接计算,通过观察方式检验结合模式,挑选出8个化合物。
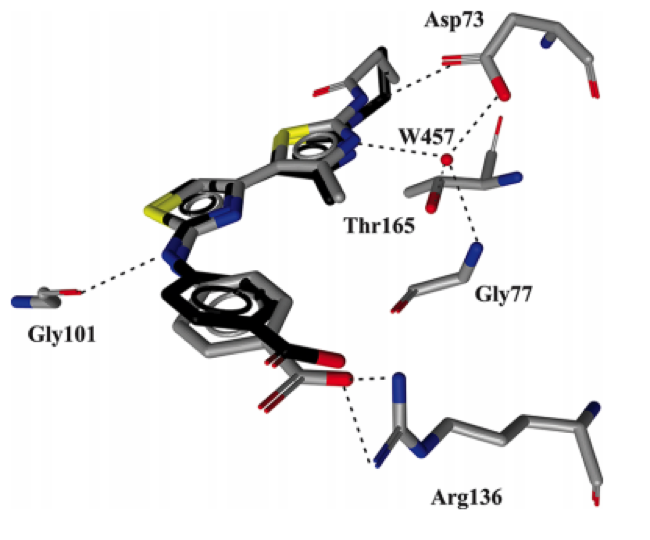
图三、GOLD计算的化合物6与G24 ATP结合位点的结合模式。
- 生物学验证
全部化合物具有DNA促旋酶抑制活性,最好的活性为1.1±0.2 μM。
- 生物物理和结构评价
分别采用DSF、SPR、MST和X-ray技术进行结合热稳性测定、分子间相互作用确证、Kd值测定和蛋白-小分子复合物结构分析。
表一、4,5’-二噻唑类化合物6和阶段二挑选的化合物18–25的E. coli DNA促旋酶抑制活性
图四、实验测定和对接得到的化合物18在ATP活性位点的结合构象。
结论
虚拟筛选、生物物理及结构生物学技术的组合运用,采用基于结构的药效团建模技术,从天然抑制剂的结构数据出发,可以发现全新骨架DNA促旋酶B型抑制剂。其中一个化合物的IC50可达1.1 ± 0.2 μM,并揭示了重要的作用位点。
亮点
- 采用两步设计策略:先以天然抑制剂的结构数据为基础进行筛选,再以第一阶段的苗头化合物建立集中库进一步筛选;
- 对药效团命中的化合物进一步进行分子对接计算;
- 采用LigandScout基于结构的药效团建模技术与GOLD分子对接方法,可发现全新骨架的抑制剂;
- 先后发现9个化合物具有低微摩尔水平的抑制活性。
- 采用多种生物物理和结构生物学技术,揭示了抑制剂的结构特征与结合模式。
关键软件与技术
LigandScout的药效团识别技术,GOLD的分子对接计算,SPR、MST、DSF及X-ray等多种生物物理和结构生物学技术。 LigandScout是奥地利InteLigand公司旗舰产品,广州墨灵格信息科技有限公司为其中国合作伙伴。